Zespół Ewolucji Funkcji Systemów Biologicznych
Czym się zajmujemy ?
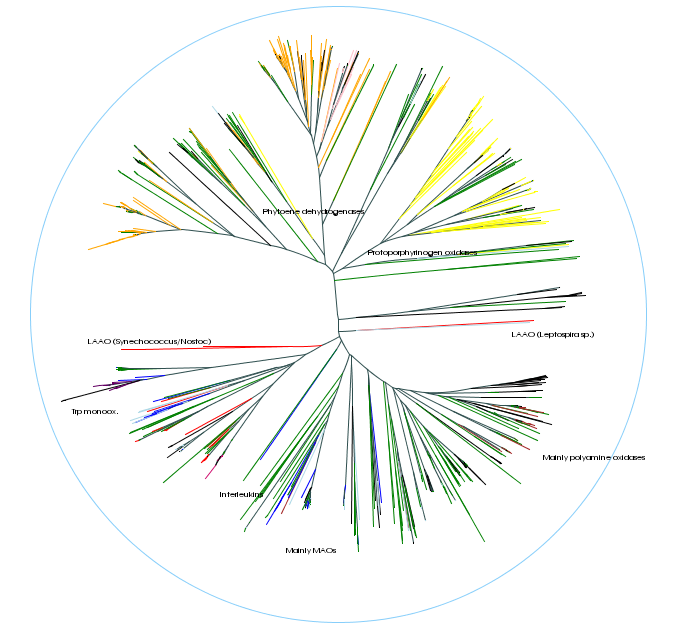
| Badamy funkcję elementów genomu (genów i sekwencji niekodujących), jako wypadkową selekcji wynikającej zarówno z ograniczeń wymaganej biologicznie funkcji, jak i z granic narzucanych żywym organizmom przez prawa natury i rachunek prawdopodobieństwa. Do analiz stosujemy warsztat z zakresu biologii molekularnej, bioinformatyki struktury i sekwencji, biofizyki oraz statystyki i teorii gier. Staramy się szukać prostych zależności w skomplikowanym świecie i tworzyć narzędzia umożliwiające jak najłatwiejsze znalezienie i zobrazowanie tych reguł. |
|
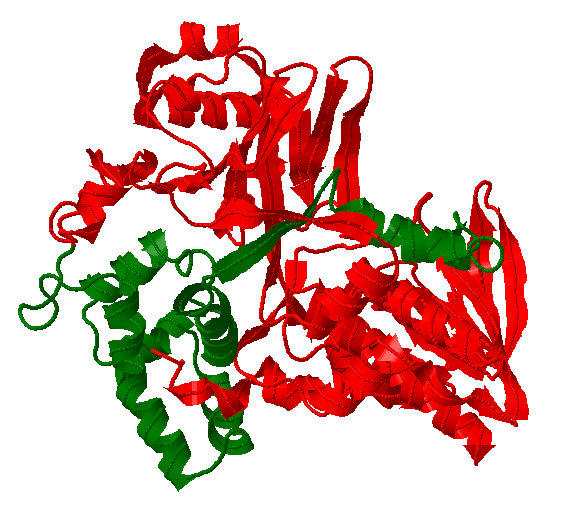 |
Aktualnie prowadzone prace dotyczą:
|
Kim jesteśmy ?
 |
 |
| Bioinformatyka: dr Grzegorz Koczyk (gkoc@igr.poznan.pl) |
Biologia molekularna: dr Adam Dawidziuk (adaw@igr.poznan.pl) dr Delfina Popiel (dpop@igr.poznan.pl) |
Nasze narzędzia i pakiety oprogramowania:
- DHcL: Domain Hierarchy and closed Loops (serwer do analizy architektury domenowej białek)
- Filogenomiczna "mapa drogowa" NR-PKSów (nieredukujących syntaz poliketydów aromatycznych) w modelowych gatunkach grzybów
Ważniejsze publikacje:
- 2015:
- Koczyk G, Dawidziuk A, Popiel D The distant siblings - a phylogenomic roadmap illuminates the origins of extant diversity in fungal aromatic polyketide biosynthesis Genome Biol Evol; 7(11):3132-3154
- Bocian A, Zwierzykowski Z, Rapacz M, Koczyk G, Ciesiolka D Metabolite profiling during cold acclimation of Lolium perenne genotypes distinct in the level of frost tolerance J Appl Genet; 56(4):439-49
- 2014:
- Popiel D, Koczyk G, Dawidziuk A, Gromadzka K, Blaszczyk L, Chelkowski J Zearalenone lactonohydrolase activity in Hypocreales and its evolutionary relationships within the epoxide hydrolase subset of a/b-hydrolases. BMC Microbiology; 14:82
- Dawidziuk A, Koczyk G, Popiel D, Kaczmarek J, Buśko M. Molecular diagnostics on the toxigenic potential of Fusarium spp. plant pathogens. J Appl Microbiol.; 116(6):1607-20
- Kroc M, Koczyk G, Święcicki W, Kilian A, Nelson MN New evidence of ancestral polyploidy in the Genistoid legume Lupinus angustifolius L. (narrow-leafed lupin) Theor Appl Genet.; 127(5):1237-49
- Stępień Ł, Koczyk G, Waśkiewicz A. Diversity of Fusarium species and mycotoxins contaminating pineapple. J Appl Genet.; 54(3):367-80.
- Dawidziuk A, Kaczmarek J, Jedryczka M. (2012) The effect of winter weather conditions on the ability of pseudothecia of Leptosphaeria maculans and L. biglobosa to release ascospores. Eur J Plant Pathol; 134(2):349-343
- Dawidziuk A, Kaczmarek J, Podlesna A, Kasprzyk I, Jedryczka M. (2012) Influence of meteorological parameters on Leptosphaeria maculans and L. biglobosa spore release in central and eastern Poland. Grana; 51(3):240-248
- Stępień L, Koczyk G, Waśkiewicz A. Genetic and phenotypic variation of Fusarium proliferatum isolates from different host species. J Appl Genet.;52(4):487-96
- Błaszczyk L, Popiel D, Chełkowski J, Koczyk G, Samuels GJ, Sobieralski K, Siwulski M. Species diversity of Trichoderma in Poland. J Appl Genet.;52(2):233-43
- Stępień L, Koczyk G, Waśkiewicz A. FUM cluster divergence in fumonisins-producing Fusarium species. Fungal Biol.;115(2):112-23
- Kaczmarek M, Koczyk G, Ziolkowski PA, Babula-Skowronska D, Sadowski J. (2009) Comparative analysis of the Brassica oleracea genetic map and the Arabidopsis thaliana genome. Genome.; 52(7):620-33
- Ziolkowski PA, Koczyk G, Galganski L, Sadowski J. (2009) Genome sequence comparison of Col and Ler lines reveals the dynamic nature of Arabidopsis chromosomes. Nucleic Acids Res.; 37(10):3189-201
- Koczyk G, Berezovsky IN. (2008) Domain Hierarchy and closed Loops (DHcL): a server for exploring hierarchy of protein domain structure. Nucleic Acids Res.; 36(Web Server issue):W239-4
- Wyrwicz LS, Koczyk G, Rychlewski L. (2008) Homologues of HSV-1 nuclear egress factor UL34 are potential phosphoinositide-binding proteins. Acta Biochim Pol.; 55(1):207-13
- Stępień Ł, Popiel D, Koczyk G, Chełkowski J. (2008) Wheat-infecting Fusarium species in Poland--their chemotypes and frequencies revealed by PCR assay. J Appl Genet.; 49(4):433-41
- Masojc B, Medrek K, Debniak T, Lubinski J, Wyrwicz LS, Koczyk G, Hoffmann M, Rychlewski L. (2007) ARLTS1 Trp149Stop mutation and the risk of ovarian cancer. Cancer Res.; 67(9):4533; author reply 4534
- Koczyk G, Wyrwicz LS, Rychlewski L. (2007) LigProf: a simple tool for in silico prediction of ligand-binding sites. J Mol Model.; 13(3):445-55
- Wyrwicz LS, Koczyk G, Rychlewski L, Plewczynski D. (2007) ProteinSplit: splitting of multi-domain proteins using prediction of ordered and disordered regions in protein sequences for virtual structural genomics. J. Phys.: Condens. Matter; 19:285222
Realizowane projekty:

